Bakteryjne systemy sekrecji
| Ten artykuł należy dopracować |
Bakteryjne systemy sekrecyjne są kompleksami białkowymi, służącymi do wydzielania substancji, obecnymi w błonach komórkowych bakterii. W szczególności są to maszyny molekularne wykorzystywane przez bakterie chorobotwórcze do wydzielania czynników wirulencji (głównie białek) w celu dokonania inwazji na komórkach gospodarza. Można je podzielić na różne typy, bazując na ich specyficznej strukturze, składzie i aktywności. Te główne różnice występują między bakteriami Gram-ujemnymi i Gram-dodatnimi. Klasyfikacja mimo to w żadnym wypadku nie jest jasna ani kompletna. Istnieje co najmniej osiem rodzajów tych kompleksów specyficznych dla bakterii Gram-ujemnych i cztery dla bakterii Gram-dodatnich, a dwa są wspólne dla obu grup.[1] Zasadniczo białka mogą być wydzielane na dwóch różnych szlakach. Jeden proces to jednoetapowy mechanizm, podczas którego białka z cytoplazmy bakterii są transportowane i dostarczane bezpośrednio przez błonę do komórki gospodarza. Kolejny z nich jest dwuetapowy. Białka są najpierw transportowane z wewnętrznej błony komórkowej, następnie osadzane w przestrzeni peryplazmatycznej, a na koniec przez zewnętrzną błonę trafiają do komórki gospodarza[2].

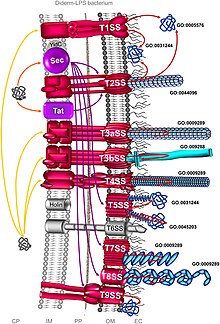
System Sec edytuj
Ogólna sekrecja (Sec) obejmuje wydzielanie niezwiniętych białek, najpierw pozostających wewnątrz komórek. U bakterii Gram-ujemnych wydzielane białko jest wysyłane do błony wewnętrznej lub przestrzeni peryplazmatycznej. W bakteriach Gram-dodatnich białko może pozostać w komórce lub jest transportowane na zewnątrz bakterii za pomocą innych systemów wydzielniczych. Wśród bakterii Gram-ujemnych niektóre takie jak Vibrio cholerae, Klebsiella pneumoniae i Yersinia enterocolitica stosują system Sec. Staphylococcus aureus i Listeria monocytogenes to bakterie Gram-dodatnie wykorzystujące system Sec[3].
System Sec wykorzystuje dwie różne ścieżki wydzielania: SecA i ścieżki cząstek rozpoznających sygnał (SRP). SecA jest ATPazowym białkiem motorycznym i ma wiele spokrewnionych ze sobą białek, takich jak SecD, SecE, SecF, SegG, SecM i SecY. SRP jest rybonukleoproteiną (kompleks białko-RNA), która rozpoznaje i kieruje określone białka do retikulum endoplazmatycznego u eukariontów lub błony komórkowej u prokariotów. Te dwa szlaki wymagają różnych białek opiekuńczych i ostatecznie wykorzystują kanał SecYEG do transportu białek przez błonę[4]. W szlaku SecA, białko SecB działa opiekuńczo, pomagając w transporcie białka do peryplazmy po jego całkowitej syntezie. W szlaku SRP YidC jest białkiem opiekuńczym transportującym białka do błony komórkowej, jeszcze w trakcie ich biosyntezy[5].
SecA/ szlak potranslacyjny edytuj
Białka są syntetyzowane na rybosomach w procesie masowego dołączania aminokwasów, zwanym translacją. W szlaku SecA opiekuńczy czynnik wyzwalający (TF) najpierw wiąże się z odsłoniętą N-końcową sekwencją sygnałową łańcucha peptydowego. W miarę wydłużania się łańcucha peptydowego TF jest zastępowany przez SecB. SecB specyficznie utrzymuje peptyd w formie niesfałdowanej i pomaga w wiązaniu czynnika SecA. Kompleks może następnie wiązać się z SecYEG, dzięki czemu SecA jest aktywowany przez wiązanie z ATP. Napędzany energią hydrolizy ATP, SecA przepycha białko przez kanał SecYEG. Kompleks SecD / F pomaga również w wyciąganiu białka z drugiej strony błony komórkowej[6].
Szlak SRP edytuj
W tym szlaku SRP współzawodniczy z TF i wiąże się z N-końcową sekwencją sygnałową. Białka błony wewnętrznej hamują proces elongacji łańcucha polipeptydowego. SRP następnie wiąże się z receptorem błonowym FtsY. Kompleks łańcucha peptydowego, SRP i FtsY jest następnie transportowany do SecY, gdzie elongacja polipeptydu jest kontynuowana[5].
System Tat edytuj
Bliźniaczy system translokacji argininy (Tat) jest podobny do Sec w procesie wydzielania białek, jednak wysyła białka tylko w formie sfałdowanej (trzeciorzędowej). Występuje u wszystkich rodzajów bakterii, a także archeonów, chloroplastów i mitochondriów roślin[7]. U bakterii układ Tat eksportuje białka z cytoplazmy przez wewnętrzną błonę komórkową; podczas gdy w chloroplastach występuje w błonie tylakoidów, gdzie wspomaga import białek ze stromy[8]. Białka Tat charakteryzują się wysoką zmiennością u różnych bakterii. Dzielimy je na trzy główne typy: TatA, TatB i TatC. Na przykład, podczas gdy u Bacillus subtilis występują tylko dwa funkcjonalne białka Tat[9], u Streptomyces coelicolor może być ich ponad sto[10]. Peptydy sygnałowe rozpoznające białka Tat charakteryzują się motywem konsensusowym Ser/Thr-Arg-Arg-X-Phe-Leu-Lys (gdzie X może być dowolnym polarnym aminokwasem). Właśnie od dwóch kolejnych arginin w tej sekwencji pochodzi nazwa bliźniaczej translokacji argininy. Zastąpienie którejkolwiek argininy prowadzi do spowolnienia lub niewydolności sekrecji[11].
Typ I edytuj
System sekrecyjny typu I (T1SS lub TOSS) występuje wśród bakterii Gram-ujemnych. Zależy to od aktywności białek opiekuńczych używających białek Hly i Tol. HlyA wiąże HlyB na błonie komórkowej, co skutkuje kaskadą sygnałową. Ta sekwencja sygnałowa jest transporterem ABC. Kompleks HlyD jest aktywowany przez kompleks HlyAB, po czym rozfałdowuje się i przemieszcza do zewnętrznej błony komórkowej. Sygnał końcowy jest rozpoznawany przez TolC w błonie wewnętrznej. HlyA jest wydzielany z błony zewnętrznej przez kanał błonowy.
T1SS transportuje przeróżne cząsteczki, w tym jony, węglowodany, leki i białka. Wydzielane związki różnią się wielkością: od małego peptydu kolicyny V u Escherichia coli mającego masę 10 kDa po białka adhezyjne LapA masie 520 kDa w komórkach Pseudomonas fluorescens[12]. Do najbardziej znanych cząsteczek podlegających sekrecji należą toksyny RTX i lipazy[czy to ważne?].
Typ II edytuj
System sekrecyjny typu II (T2SS) zależy od systemu Sec lub Tat dla początkowej fazy sekrecji wewnątrz komórki bakteryjnej. Z przestrzeni peryplazmatycznej białka są wydzielane przez błonę zewnętrzną. Sekretyny są multimerycznymi (12–14 podjednostek) kompleksami białek porotwórczych. Sekretyna jest wspierana przez 10-15 innych białek błony wewnętrznej i zewnętrznej, tworząc razem kompletny aparat sekrecyjny[13].
Typ III edytuj
System sekrecyjny typu III (T3SS lub TTSS) jest strukturalnie podobny i spokrewniony z ciałkiem podstawowym wici bakteryjnej. Znaleziony u w niektórych najbardziej zjadliwych bakterii Gram-ujemnych, takich jak Salmonella, Shigella, Yersinia, Vibrio, jest używany do transportu toksycznych białek do komórek eukariotycznych. Ze względu na swoją strukturę jest często opisywany jako injektosom. Po odkryciu go u Yersinia pestis, stwierdzono, że T3SS może wstrzykiwać toksyny bezpośrednio z cytoplazmy bakteryjnej do wnętrza komórek gospodarza[14].
Typ IV edytuj
Systemy sekrecyjne typu IV (T4SS lub TFSS) jest spokrewniony z bakteryjnym systemem koniugacji, za pomocą którego różne bakterie mogą wymieniać się DNA. Uczestniczące w tej aktywności bakterie mogą należeć do tego samego lub różnych gatunków bakterii Gram-ujemnych. System może transportować pojedyncze białka, ale również kompleksy białko-białko i DNA-białko. Wydzielane białko jest przenoszone bezpośrednio z komórki biorcy przez błony komórkowe. Agrobacterium tumefaciens, z którego układ ten został po raz pierwszy wyizolowany, wykorzystuje ten system do wysyłania części T-DNA plazmidu Ti do komórek roślinnych, w wyniku czego powstaje galas (guz). Helicobacter pylori wykorzystuje go, aby dostarczać CagA do komórek nabłonka żołądka, co skutkuje kancerogenezą i rozwojem raka żołądka[15]. Bordetella pertussis, bakteria wywołująca krztusiec, wydziela toksynę krztuścową częściowo przez T4SS. Legionella pneumophila powodująca legionelozę ma T4SS zwany icm/dot (intra cellular multiplication/defect in organelle trafficking genes), który transportuje wiele białek bakteryjnych do eukariotycznego hosta[16]. Niedawno wykazano[kiedy?], że patogen roślinny Xanthomonas citri wykorzystuje swój T4SS do wydzielania efektorów, które są śmiertelne dla innych gatunków bakterii, co czyni ten system głównym wyznacznikiem sprawności międzygatunkowej konkurencji bakteryjnej[17][18]. Prototypowym[co to znaczy] układem wydzielniczym typu IVA jest kompleks VirB Agrobacterium tumefaciens[19].
Typ V edytuj
Systemy sekrecyjne typu V (T5SS) różnią się od innych systemów tego samego rodzaju tym, że wydzielają się same i angażują jedynie zewnętrzną błonę komórkową. Aby wydzielane białko mogło przejść przez wewnętrzną błonę komórkową, T5SS jest zależne od systemu Sec. T5SS posiadają domenę β-beczułki, która włącza się do zewnętrznej błony komórkowej i tworzy kanał transportujący wydzielane białko. W tym procesie nazywane są również systemami autotransporterów[20]. Kiedy wydzielane białka są odsłonięte na zewnątrz, autotransportery są odcinane (rozszczepiane), uwalniając białko z domeny β-beczki. Przykładem autotransportera jest Trimeric Autotransporter Adhesins[21].
Typ VI edytuj
Systemy sekrecyjne typu VI (T6SS) zostały wyizolowane z Vibrio cholerae i Pseudomonas aeruginosa przez zespół Johna Mekalanosa w Harvard Medical School w 2006 roku[22][23]. Zostały odkryte, gdy mutacje w genach Vibrio Cholerae Hcp i VrgG zmniejszyły wirulencję i patogenność bakterii[24][25]. Oprócz swojej głównej funkcji czynnika chorobotwórczego, T6SS są również zaangażowane w obronę przed prostymi eukariotycznymi drapieżnikami[czyli jakimi] oraz w interakcje międzybakteryjne[26][27]. Gen T6SS tworzy klaster genów składający się z ponad 15 genów. Geny Hcp i VgrG są najbardziej uniwersalne. Strukturalne podobieństwo T6SS do peplomeru ogona faga T4 sugeruje, że proces infekcji jest podobny do wnikania faga do komórki[28].
Typ VII edytuj
System sekrecyjny typu VII (T7SS) jest obecny w bakteriach Gram-dodatnich i mykobakteriach, takich jak M. tuberculosis, M. bovis, Streptomyces coelicolor i S. aureus. u Bacillus subtilis i S. aureus jest również nazywany systemem T7b. Jest złożony z dwóch podstawowych składników: związanej z błoną heksamerycznej ATPazy należącej do rodziny białek FtsK/SpoIIIE[29] oraz jednego z białek EsxA/EsxB, takiego jak EsaA, EsaD, EsxB, EsxD, jak również system Ess (EssA, EssB i EsxC znalezione w S. aureus)[30]. EsxA i EsxB należą do nadrodziny białek WXG100, które tworzą struktury dimerycznych, helikalnych szpilek do włosów. S. aureus T7SS wydziela duże toksyny zwane EsaD należące do rodziny nukleaz. EsaD staje się nieszkodliwa podczas biosyntezy prowadząnej z pomocą jej antytoksyny EsaG. Kompleks EsaD-EsaG wiąże się następnie z EsaE. Część EsaE wiąże się z EssC, który jest ATPazą kompleksu T7SS. Podczas sekrecji EsaG pozostaje w cytoplazmie, a jedynie EsaD i EsaE są razem wydzielane. W niektórych szczepach S. aureus EsaD nie jest wytwarzana, ale zamiast tego powstają dwie kopie białek podobnych do EsaG. Może to wyjaśniać występowanie T7SS u niepatogennych gatunków, takich jak B. subtilis i S. coelicolor[31].
Typ IX edytuj
Systemy sekrecyjne typu IX (T9SS) występują u bakteryjnej linii Fibrobacteres-Chlorobi-Bacteroidetes, której gatunki składowe posiadają błonę zewnętrzną. System jest różnie zaangażowany w jednym typie ruchu ślizgowego, podczas właściwego kierowania pewnych czynników wirulencji na powierzchnię komórki oraz w degradacji kompleksu biopolimerów[32]. T9SS jest również znany jako wydzielina Por, po ustnym patogenie Porphyromonas gingivalis. Opisano co najmniej szesnaście elementów strukturalnych tego systemu, w tym PorU, transpeptydazę sortującą białka, która usuwa C-końcowy sygnał sortowania z białek cargo i pośredniczy w ich przyłączaniu do lipopolisacharydu.
Przypisy edytuj
- ↑ Erin R. Green, Joan Mecsas, Bacterial Secretion Systems: An Overview, „Microbiology Spectrum”, 4 (1), 2016, DOI: 10.1128/microbiolspec.VMBF-0012-2015, ISSN 2165-0497, PMID: 26999395, PMCID: PMC4804464 [dostęp 2024-03-31].
- ↑ Katarzyna M. Bocian-Ostrzycka i inni, Bacterial thiol oxidoreductases — from basic research to new antibacterial strategies, „Applied Microbiology and Biotechnology”, 101 (10), 2017, s. 3977–3989, DOI: 10.1007/s00253-017-8291-8, ISSN 0175-7598, PMID: 28409380, PMCID: PMC5403849 [dostęp 2024-03-31] (ang.).
- ↑ Barbara A. Bensing i inni, Selective transport by SecA2: An expanding family of customized motor proteins, „Biochimica et Biophysica Acta (BBA) - Molecular Cell Research”, 1843 (8), 2014, s. 1674–1686, DOI: 10.1016/j.bbamcr.2013.10.019, PMID: 24184206, PMCID: PMC4007388 [dostęp 2024-03-31] (ang.).
- ↑ Jennine M. Crane, Linda L. Randall, The Sec System: Protein Export in Escherichia coli, Susan T. Lovett, Harris D. Bernstein (red.), „EcoSal Plus”, 7 (2), 2017, DOI: 10.1128/ecosalplus.esp-0002-2017, ISSN 2324-6200, PMID: 29165233, PMCID: PMC5807066 [dostęp 2024-03-31] (ang.).
- ↑ a b Lu Zhu, H. Ronald Kaback, Ross E. Dalbey, YidC Protein, a Molecular Chaperone for LacY Protein Folding via the SecYEG Protein Machinery, „Journal of Biological Chemistry”, 288 (39), 2013, s. 28180–28194, DOI: 10.1074/jbc.M113.491613, PMID: 23928306, PMCID: PMC3784728 [dostęp 2024-03-31] (ang.).
- ↑ Jelger A. Lycklama a Nijeholt, Arnold J.M. Driessen, The bacterial Sec-translocase: structure and mechanism, „Philosophical Transactions of the Royal Society B: Biological Sciences”, 367 (1592), 2012, s. 1016–1028, DOI: 10.1098/rstb.2011.0201, PMID: 22411975, PMCID: PMC3297432 [dostęp 2024-03-31] (ang.).
- ↑ Ming-Ren Yen i inni, equence and phylogenetic analyses of the twin-arginine targeting (Tat) protein export system, „Archives of Microbiology”, 177 (6), 2002, s. 441–450, DOI: 10.1007/s00203-002-0408-4, PMID: 12029389 [dostęp 2024-03-31].
- ↑ Philip A. Lee, Danielle Tullman-Ercek, George Georgiou, The Bacterial Twin-Arginine Translocation Pathway, „Annual Review of Microbiology”, 60 (1), 2006, s. 373–395, DOI: 10.1146/annurev.micro.60.080805.142212, PMID: 16756481, PMCID: PMC2654714 [dostęp 2024-03-31] (ang.).
- ↑ Jan D.H. Jongbloed i inni, Two minimal Tat translocases in Bacillus, „Molecular Microbiology”, 54 (5), 2004, s. 1319–1325, DOI: 10.1111/j.1365-2958.2004.04341.x, PMID: 15554971 [dostęp 2024-03-31] (ang.).
- ↑ Haiming Li i inni, Determining the functionality of putative Tat-dependent signal peptides in Streptomyces coelicolor A3(2) by using two different reporter proteins, „Microbiology”, 151 (7), 2005, s. 2189–2198, DOI: 10.1099/mic.0.27893-0, PMID: 16000709 [dostęp 2024-03-31] (ang.).
- ↑ N.R. Stanley, T. Palmer, B.C. Berks, The twin arginine consensus motif of Tat signal peptides is involved in Sec-independent protein targeting in Escherichia coli, „Journal of Biological Chemistry”, 275 (16), 2000, s. 11591–11596, DOI: 10.1074/jbc.275.16.11591, ISSN 0021-9258, PMID: 10766774 [dostęp 2024-03-31].
- ↑ Chelsea D. Boyd i inni, Structural features of the Pseudomonas fluorescens biofilm adhesin LapA required for LapG-dependent cleavage, biofilm formation, and cell surface localization, „Journal of Bacteriology”, 196 (15), 2014, s. 2775–2788, DOI: 10.1128/JB.01629-14, ISSN 1098-5530, PMID: 24837291, PMCID: PMC4135675 [dostęp 2024-03-31].
- ↑ Konstantin V. Korotkov, Maria Sandkvist, Wim G.J. Hol, The type II secretion system: biogenesis, molecular architecture and mechanism, „Nature Reviews. Microbiology”, 10 (5), 2012, s. 336–351, DOI: 10.1038/nrmicro2762, ISSN 1740-1534, PMID: 22466878, PMCID: PMC3705712 [dostęp 2024-03-31].
- ↑ Daniela Büttner, Protein export according to schedule: architecture, assembly, and regulation of type III secretion systems from plant- and animal-pathogenic bacteria, „Microbiology and molecular biology reviews: MMBR”, 76 (2), 2012, s. 262–310, DOI: 10.1128/MMBR.05017-11, ISSN 1098-5557, PMID: 22688814, PMCID: PMC3372255 [dostęp 2024-03-31].
- ↑ Masanori Hatakeyama, Hideaki Higashi, Helicobacter pylori CagA: a new paradigm for bacterial carcinogenesis, „Cancer Science”, 96 (12), 2005, s. 835–843, DOI: 10.1111/j.1349-7006.2005.00130.x, ISSN 1347-9032, PMID: 16367902 [dostęp 2024-03-31].
- ↑ Eric Cascales, Peter J. Christie, The versatile bacterial type IV secretion systems, „Nature Reviews. Microbiology”, 1 (2), 2003, s. 137–149, DOI: 10.1038/nrmicro753, ISSN 1740-1526, PMID: 15035043, PMCID: PMC3873781 [dostęp 2024-03-31].
- ↑ Diorge P. Souza i inni, Bacterial killing via a type IV secretion system, „Nature Communications”, 6, 2015, s. 6453, DOI: 10.1038/ncomms7453, ISSN 2041-1723, PMID: 25743609 [dostęp 2024-03-31] (ang.).
- ↑ Germán G. Sgro i inni, Cryo-EM structure of the bacteria-killing type IV secretion system core complex from Xanthomonas citri, „Nature Microbiology”, 3 (12), 2018, s. 1429–1440, DOI: 10.1038/s41564-018-0262-z, ISSN 2058-5276, PMID: 30349081, PMCID: PMC6264810 [dostęp 2024-03-31] (ang.).
- ↑ Peter J. Christie i inni, Biogenesis, architecture, and function of bacterial type IV secretion systems, „Annual Review of Microbiology”, 59, 2005, s. 451–485, DOI: 10.1146/annurev.micro.58.030603.123630, ISSN 0066-4227, PMID: 16153176, PMCID: PMC3872966 [dostęp 2024-03-31].
- ↑ David G. Thanassi i inni, Protein secretion in the absence of ATP: the autotransporter, two-partner secretion and chaperone/usher pathways of gram-negative bacteria (review), „Molecular Membrane Biology”, 22 (1-2), 2005, s. 63–72, DOI: 10.1080/09687860500063290, ISSN 0968-7688, PMID: 16092525 [dostęp 2024-03-31].
- ↑ R Gerlach, M Hensel, Protein secretion systems and adhesins: The molecular armory of Gram-negative pathogens, „International Journal of Medical Microbiology”, 297 (6), 2007, s. 401–415, DOI: 10.1016/j.ijmm.2007.03.017, PMID: 17482513 [dostęp 2024-03-31] (ang.).
- ↑ Stefan Pukatzki i inni, Identification of a conserved bacterial protein secretion system in Vibrio cholerae using the Dictyostelium host model system, „Proceedings of the National Academy of Sciences of the United States of America”, 103 (5), 2006, s. 1528–1533, DOI: 10.1073/pnas.0510322103, ISSN 0027-8424, PMID: 16432199, PMCID: PMC1345711, Bibcode: 2006PNAS..103.1528P, JSTOR: 30048406 [dostęp 2024-03-31].
- ↑ Joseph D. Mougous i inni, A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus, „Science”, 312 (5779), 2006, s. 1526–1530, DOI: 10.1126/science.1128393, ISSN 1095-9203, PMID: 16763151, PMCID: PMC2800167 [dostęp 2024-03-31].
- ↑ Lewis Eh Bingle, Christopher M. Bailey, Mark J. Pallen, Type VI secretion: a beginner's guide, „Current Opinion in Microbiology”, 11 (1), 2008, s. 3–8, DOI: 10.1016/j.mib.2008.01.006, ISSN 1369-5274, PMID: 18289922 [dostęp 2024-03-31].
- ↑ Eric Cascales, The type VI secretion toolkit, „EMBO reports”, 9 (8), 2008, s. 735–741, DOI: 10.1038/embor.2008.131, ISSN 1469-3178, PMID: 18617888, PMCID: PMC2515208 [dostęp 2024-03-31].
- ↑ Sandra Schwarz, Rachel D. Hood, Joseph D. Mougous, What is type VI secretion doing in all those bugs?, „Trends in Microbiology”, 18 (12), 2010, s. 531–537, DOI: 10.1016/j.tim.2010.09.001, ISSN 1878-4380, PMID: 20961764, PMCID: PMC2991376 [dostęp 2024-03-31].
- ↑ Sarah J. Coulthurst, The Type VI secretion system - a widespread and versatile cell targeting system, „Research in Microbiology”, 164 (6), 2013, s. 640–654, DOI: 10.1016/j.resmic.2013.03.017, ISSN 1769-7123, PMID: 23542428 [dostęp 2024-03-31].
- ↑ Julie M. Silverman i inni, Structure and regulation of the type VI secretion system, „Annual Review of Microbiology”, 66, 2012, s. 453–472, DOI: 10.1146/annurev-micro-121809-151619, ISSN 1545-3251, PMID: 22746332, PMCID: PMC3595004 [dostęp 2024-03-31].
- ↑ Khaled A. Aly i inni, Isolation of a Membrane Protein Complex for Type VII Secretion in Staphylococcus aureus, „Journal of Bacteriology”, 199 (23), 2017, e00482–17, DOI: 10.1128/JB.00482-17, PMID: 28874412, PMCID: PMC5686593 [dostęp 2024-04-01].
- ↑ Holger Kneuper i inni, Heterogeneity in ess transcriptional organization and variable contribution of the Ess/Type VII protein secretion system to virulence across closely related Staphylocccus aureus strains, „Molecular Microbiology”, 93 (5), 2014, s. 928–943, DOI: 10.1111/mmi.12707, PMID: 25040609, PMCID: PMC4285178 [dostęp 2024-04-01].
- ↑ Zhenping Cao i inni, The type VII secretion system of Staphylococcus aureus secretes a nuclease toxin that targets competitor bacteria, „Nature Microbiology”, 2, 2016, s. 16183, DOI: 10.1038/nmicrobiol.2016.183, PMID: 27723728, PMCID: PMC5325307 [dostęp 2024-04-01].
- ↑ Paul D. Veith i inni, Type IX secretion: the generation of bacterial cell surface coatings involved in virulence, gliding motility and the degradation of complex biopolymers, „Molecular Microbiology”, 106 (1), 2017, s. 35–53, DOI: 10.1111/mmi.13752, PMID: 28714554 [dostęp 2024-04-01].