Laktaza
Laktaza – enzym katalizujący hydrolizę disacharydu laktozy na jej dwa komponenty – glukozę i galaktozę. Wytwarzana jest przez zwierzęta, rośliny, grzyby, bakterie i drożdże. W zależności od mechanizmu działania laktazy klasyfikowane są jako β-D-galaktozydazy (EC 3.2.1.23[1]) lub β-D-glukozydazy (EC 3.2.1.21[2])[3].

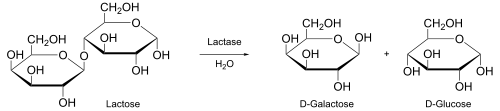
.
Laktaza ssacza
edytujLaktaza ssacza (β-D-galaktozydaza[3], tilaktaza[4]) ma dodatkową aktywność hydrolazy floryzyny[5]. Wytwarzana jest w rąbku szczoteczkowym nabłonka jelita cienkiego[6].
Laktaza ludzka
edytujJest niezbędna w procesie trawienia laktozy zawartej w mleku, zarówno ludzkim, jak i krowim (w którym jej zawartość wynosi 4,8%)[3]. U części ludzi, w wieku 2–12 lat ustaje jej wytwarzanie[6]. W efekcie dochodzi do fermentacji laktozy przez florę bakteryjną jelita[3], co – często, choć nie zawsze[6] – powoduje bóle brzucha, biegunkę, wzdęcia i inne objawy żołądkowo-jelitowe, określane łącznie jako nietolerancja laktozy[3].
Właściwości
edytujLaktaza ludzka osiąga maksymalną aktywność w temperaturze ok. 48 °C i pH równym 6,5. Gen laktazy jest zlokalizowany w drugim chromosomie (2q21).
Genetyka
edytujUtrzymanie wydzielania laktazy u dorosłych ma podłoże genetyczne i jest różne u różnych populacji. Najczęściej występuje w północnej Europie, u niektórych ludów Afryki, nomadów, którzy od dawna pili słodkie mleko.
Cecha ta nie jest spowodowana mutacjami w obrębie samego genu laktazy LCT, lecz mutacjami w obszarach intronów genu MCM6, które wpływają na ekspresję genu LCT. W Europie za tolerancję laktozy odpowiadają dwie mutacje: przede wszystkim zmiana C na T w pozycji −13910, i nieco rzadsza zmiana G na A w pozycji −22018. W Afryce Wschodniej z tolerancją na laktozę skorelowane są inne polimorfizmy, również znajdujące się w intronach genu MCM6.
Uważa się, że allele niosące utrzymującą się u dorosłych tolerancję na laktozę były rzadkie, a rozpowszechniły się dopiero wtedy, kiedy ludzie udomowili bydło i zaczęli korzystać z jego mleka. Badania próbek neolitycznych kości ludzkich z terenów Europy sugerują, że wtedy ta mutacja nie była rozpowszechniona[7].
Zastosowanie przemysłowe
edytujLaktaza na skalę przemysłową jest produkowana z grzybów z rodzaju Aspergillus i drożdży z rodzaju Kluyveromyces. Enzym pochodzący z grzybów jest aktywny w środowisku kwaśnym i jest stosowany przy przerobie serwatki, natomiast produkt wytwarzany przez drożdże ma maksimum aktywności przy pH ok. 7 i jest wykorzystywany do produkcji mleka bezlaktozowego[3].
Laktaza jest także stosowana przy produkcji lodów. Ponieważ glukoza i galaktoza są słodsze niż laktoza, laktaza daje wrażenie lepszego smaku, poza tym obniżenie zawartości laktozy nadaje lodom lepszą płynność i gładszą jednolitą strukturę.[potrzebny przypis]
Przypisy
edytuj- ↑ EC 3.2.1.23 [online], www.sbcs.qmul.ac.uk [dostęp 2018-03-24] (ang.).
- ↑ EC 3.2.1.21 [online], www.sbcs.qmul.ac.uk [dostęp 2018-03-24] (ang.).
- ↑ a b c d e f Bin Li i inni, Preparation of lactose-free pasteurized milk with a recombinant thermostable β-glucosidase from Pyrococcus furiosus, „BMC biotechnology”, 13, 2013, s. 73, DOI: 10.1186/1472-6750-13-73, PMID: 24053641, PMCID: PMC4016594 (ang.).
- ↑ Tilactase [online], DrugBank [dostęp 2018-10-05] (ang.).
- ↑ N. Mantei i inni, Complete primary structure of human and rabbit lactase-phlorizin hydrolase: implications for biosynthesis, membrane anchoring and evolution of the enzyme, „The EMBO journal”, 7 (9), 1988, s. 2705–2713, PMID: 2460343, PMCID: PMC457059 (ang.).
- ↑ a b c Rejane Mattar, Daniel Ferraz de Campos Mazo, Flair José Carrilho, Lactose intolerance: diagnosis, genetic, and clinical factors, „Clinical and Experimental Gastroenterology”, 5, 2012, s. 113–121, DOI: 10.2147/CEG.S32368, PMID: 22826639, PMCID: PMC3401057 (ang.).
- ↑ J. Burger i inni, Absence of the lactase-persistence-associated allele in early Neolithic Europeans, „Proceedings of the National Academy of Sciences of the United States of America”, 104 (10), 2007, s. 3736–3741, DOI: 10.1073/pnas.0607187104, PMID: 17360422, PMCID: PMC1820653 (ang.).